Dopo le scoperte di Morgan, in molti cercarono di stabilire se fosse il DNA o le proteine, il materiale genetico dei cromosomi.
Nel 1928 Griffith studiando i batteri scoprì il fenomeno della trasformazione, una variazione del genotipo e del fenotipo legata all'assimilazione di DNA estraneo da parte di una cellula.
Avery nel 1944 sostenne che l'agente trasformate era il DNA, ma la sua scoperta fu accolta con scetticismo, dato che molti credevano che fossero le proteine il materiale genetico per eccellenza.
Nel 1952 Hershey e Chase studiando i batteriofagi sostennero che era il DNA il materiale genetico del batterio T2, che era capace di riprogrammare la cellula per fargli produrre altri virus, e provarono la loro teoria grazie all'uso di isotopi radiottivi per marcare il DNA, dimostrando così che le proteine non entravano in gioco in questo processo di trasmissione dei geni.
Altre prove sono legate alla distribuzione di DNA durante la mitosi, e Chargaff nel 1947 dimostrò la composizione del DNA (la base azotata è cmq sempre uguale: Adenina A, Timina T, Guanina G, Citosina C) varia da una specie ad un'altra.
Inoltre Chargaff scoprì le uguaglianze A=T e G=C comuni a tutte le molecole di DNA, adesso note come regole di Chargaff.
La prova finale fu' data da Watson e Crick che nel 1953 scoprirono la struttura a doppia elica del DNA grazie ad una cristallografia a raggi x della studiosa Rosalind Franklin.
Questi studiosi scoprirono che l'elica del DNA compie un giro completo ogni 3,4nm nel senso della sua lunghezza, e che le basi azotate erano appaiate in modo che l'adenina stava con la timina e la guanina con la citosina.
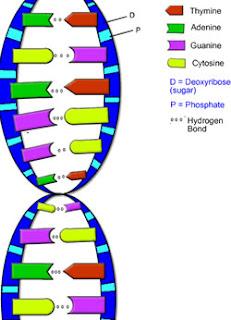
L'adenina e la guanina sono purine, basi azotate con 2 anelli organici, mentre la citosina e la timina sono pirimidine, basi con un solo anello.
Quindi l'adenina forma 2 legami ad idrogeno solo con la timina e la guanina forma 3 legami ad idrogeno solo con la citosina.
La quantità di guanina è quindi uguale a quella di citosina e quella di adenina a quella di timina, inoltre la sequenza lineare delle 4 basi può essere variata in modo infinito, ed ogni gene possiede un unico ordine o sequenza di basi.
Il modello semiconservativo di Watson e Crick prevede che quando la doppia elica si duplica, ciascuna delle molecole figlie è costituita da una parte della vecchia molecola genitrice ed una nuova parte.
Ognuna delle cellule umane ha circa 6 miliardi di paia di basi, ciò nonostante una cellua impiega poche ore per copiare tutto il suo DNA e questa replicazione è effettuata con pochissimi errori, circa uno ogni miliardo di nucleotidi.
La replicazione del DNA inizia in siti detti origini di replicazione, dove le proteine si legano al DNA separando i 2 filamenti e aprendo una bolla di replicazione.
La replicazione procede in entrambe le direzioni fino a che non è stata copiata l'intera molecola.
All'estremità di una bolla di replicazione c'è una forca di replicazione, una regione a forma di Y in cui i nuovi filamenti di DNA si allungano.
Quest'allungamento è catalizzato dagli enzimi DNA polimerasi che lega i nucleotidi appaiati alle basi all'estremità in accrescimento del nuovo filamento, ad una velocità di circa 50 nucleotidi al secondo nelle cellule umane.
La DNA polimerasi aggiunge nucleotidi nell'estremità 3' libera di un filamento crescente di DNA ma non nell'estremità 5' e quindi un nuovo filamento di DNA può allungarsi solo in direzione 5' -> 3'.
La polimerasi si posiziona nella forca di replicazione di un filamento stampo e aggiunge continuamente nucleotidi al filamento complementare man mano che la forca si estende e il filamento formato con questo meccanismo viene detto filamento guida.
Per allungare l'altro nuovo filamento la polimerasi deve lavorare sull'altro stampo nella direzione che si allontana dalla forca, e il DNA sintetizzato in questa direzione è detto filamento ritardato.
La molecola di polimerasi si allontana dalla bolla di replicazione quando questa si apre, e replica un breve segmento di DNA.
Il filamento ritardato è sintetizzato inizialmente come un insieme di brevi segmenti detti frammenti di Okazaki.
L'enzima DNA ligasi unisce gli scheletri zucchero-fosfato dei frammenti di Okazaki per generare un singolo filamento di DNA.
La DNA polimerasi non può iniziare la sintesi di un filamento polinucleotidico, può solo aggiungere nucleotidi all'estremità 3' di un filamento preesistente. L'innesco è rappresentato da un breve tratto di RNA sintetizzato dall'enzima primasi, ed ogni innesco è alla fine sostituito dal DNA.
Invece nel filamento ritardato ogni singolo frammento di Okazaki deve essere innescato e questi inneschi sono convertiti in DNA prima che la ligasi unisca assieme i frammenti.
L'elicasi è un'enzima che svolge la doppia elica a livello della forca di replicazione, separando i 2 filamenti.
La proteina che lega il DNA a filamento singolo si allinea lungo i filamenti di DNA spaiati, tenendoli separati, mentre questi funzionano come stampi per la sintesi dei nuovi filamenti complementari.
Enzimi che controllano e correggono gli errori nella replicazione del DNA
Gli errori nell'appaiamento iniziale tra i nuovi nucleotidi e quelli del filamento stampo sono abbastanza frequenti.
La polimerasi stessa effettua un controllo degli errori durante la replicazione, quando trova un nucleotide appaiato in modo improprio, lo rimuove e riprende la sintesi.
Per la riparazione degli appaiamenti scorretti sfuggiti alla polimerasi, le cellule impiegano enzimi speciali che riconoscono e correggono i nucleotidi appaiati impropriamente.
La nucleasi è l'enzima che che rimuove la porzione danneggiata, poi sostituita da altri nucleotidi, grazie alla polimerasi e la ligasi, in un processo chiamato riparazione per escissione dei nucleotidi.
La malattia ereditaria xeroderma pigmentoso è causata da un deficit di un enzima implicato nella riparazione per escissione, e comporta ipersensibilità alla luce e tumori alla pelle.
In linea di massima, la maggior parte dei processi di riparazione prevede l'intervento di molecole di DNA polimerasi.
Il normale replicamento di DNA non permette di completare l'estremità 5', e quindi cicli ripetuti di replicazione porterebbero alla produzione di molecole di DNA sempre più corte.
I telomeri sono speciali sequenze nucleotidiche, TTAGGGG, che non contengono geni.
Il DNA telomerico protegge i geni dell'organismo dall'erosione ed impedisce che le estremità attivino sistemi cellulari per monitorare il danno a carico del DNA.
La telomerasi è un particolare enzima che consente di ripristinare i telomeri accorciati, catalizzando il loro allungamento, grazie ad una molecola RNA che agisce come stampo per i nuovi segmenti del telomero all'estremità 3'.
La telomerasi non è presente nella maggior parte delle cellule pluricellulari, e quindi nelle cellule somatiche il DNA tende ad essere più corto nelle cellule degli anziani.
Quindi è probabile che i telomeri siano la causa della limitazione della durata della vita degli organismi.
La telomerasi è presente nelle cellule germinali che danno origine ai gamenti e quindi è presente nei neonati.
Purtroppo la telomerasi mantiene vive le cellule del cancro non facendo accorciare i telomeri.
Nel 1928 Griffith studiando i batteri scoprì il fenomeno della trasformazione, una variazione del genotipo e del fenotipo legata all'assimilazione di DNA estraneo da parte di una cellula.
Avery nel 1944 sostenne che l'agente trasformate era il DNA, ma la sua scoperta fu accolta con scetticismo, dato che molti credevano che fossero le proteine il materiale genetico per eccellenza.
Nel 1952 Hershey e Chase studiando i batteriofagi sostennero che era il DNA il materiale genetico del batterio T2, che era capace di riprogrammare la cellula per fargli produrre altri virus, e provarono la loro teoria grazie all'uso di isotopi radiottivi per marcare il DNA, dimostrando così che le proteine non entravano in gioco in questo processo di trasmissione dei geni.
Altre prove sono legate alla distribuzione di DNA durante la mitosi, e Chargaff nel 1947 dimostrò la composizione del DNA (la base azotata è cmq sempre uguale: Adenina A, Timina T, Guanina G, Citosina C) varia da una specie ad un'altra.
Inoltre Chargaff scoprì le uguaglianze A=T e G=C comuni a tutte le molecole di DNA, adesso note come regole di Chargaff.
La prova finale fu' data da Watson e Crick che nel 1953 scoprirono la struttura a doppia elica del DNA grazie ad una cristallografia a raggi x della studiosa Rosalind Franklin.
Questi studiosi scoprirono che l'elica del DNA compie un giro completo ogni 3,4nm nel senso della sua lunghezza, e che le basi azotate erano appaiate in modo che l'adenina stava con la timina e la guanina con la citosina.
L'adenina e la guanina sono purine, basi azotate con 2 anelli organici, mentre la citosina e la timina sono pirimidine, basi con un solo anello.
Quindi l'adenina forma 2 legami ad idrogeno solo con la timina e la guanina forma 3 legami ad idrogeno solo con la citosina.
La quantità di guanina è quindi uguale a quella di citosina e quella di adenina a quella di timina, inoltre la sequenza lineare delle 4 basi può essere variata in modo infinito, ed ogni gene possiede un unico ordine o sequenza di basi.
Replicazione e riparazione del DNA
Il modello semiconservativo di Watson e Crick prevede che quando la doppia elica si duplica, ciascuna delle molecole figlie è costituita da una parte della vecchia molecola genitrice ed una nuova parte.
Ognuna delle cellule umane ha circa 6 miliardi di paia di basi, ciò nonostante una cellua impiega poche ore per copiare tutto il suo DNA e questa replicazione è effettuata con pochissimi errori, circa uno ogni miliardo di nucleotidi.
La replicazione del DNA inizia in siti detti origini di replicazione, dove le proteine si legano al DNA separando i 2 filamenti e aprendo una bolla di replicazione.
La replicazione procede in entrambe le direzioni fino a che non è stata copiata l'intera molecola.
All'estremità di una bolla di replicazione c'è una forca di replicazione, una regione a forma di Y in cui i nuovi filamenti di DNA si allungano.
Quest'allungamento è catalizzato dagli enzimi DNA polimerasi che lega i nucleotidi appaiati alle basi all'estremità in accrescimento del nuovo filamento, ad una velocità di circa 50 nucleotidi al secondo nelle cellule umane.
La DNA polimerasi aggiunge nucleotidi nell'estremità 3' libera di un filamento crescente di DNA ma non nell'estremità 5' e quindi un nuovo filamento di DNA può allungarsi solo in direzione 5' -> 3'.
La polimerasi si posiziona nella forca di replicazione di un filamento stampo e aggiunge continuamente nucleotidi al filamento complementare man mano che la forca si estende e il filamento formato con questo meccanismo viene detto filamento guida.
Per allungare l'altro nuovo filamento la polimerasi deve lavorare sull'altro stampo nella direzione che si allontana dalla forca, e il DNA sintetizzato in questa direzione è detto filamento ritardato.
La molecola di polimerasi si allontana dalla bolla di replicazione quando questa si apre, e replica un breve segmento di DNA.
Il filamento ritardato è sintetizzato inizialmente come un insieme di brevi segmenti detti frammenti di Okazaki.
L'enzima DNA ligasi unisce gli scheletri zucchero-fosfato dei frammenti di Okazaki per generare un singolo filamento di DNA.
La DNA polimerasi non può iniziare la sintesi di un filamento polinucleotidico, può solo aggiungere nucleotidi all'estremità 3' di un filamento preesistente. L'innesco è rappresentato da un breve tratto di RNA sintetizzato dall'enzima primasi, ed ogni innesco è alla fine sostituito dal DNA.
Invece nel filamento ritardato ogni singolo frammento di Okazaki deve essere innescato e questi inneschi sono convertiti in DNA prima che la ligasi unisca assieme i frammenti.
L'elicasi è un'enzima che svolge la doppia elica a livello della forca di replicazione, separando i 2 filamenti.
La proteina che lega il DNA a filamento singolo si allinea lungo i filamenti di DNA spaiati, tenendoli separati, mentre questi funzionano come stampi per la sintesi dei nuovi filamenti complementari.
Enzimi che controllano e correggono gli errori nella replicazione del DNA
Gli errori nell'appaiamento iniziale tra i nuovi nucleotidi e quelli del filamento stampo sono abbastanza frequenti.
La polimerasi stessa effettua un controllo degli errori durante la replicazione, quando trova un nucleotide appaiato in modo improprio, lo rimuove e riprende la sintesi.
Per la riparazione degli appaiamenti scorretti sfuggiti alla polimerasi, le cellule impiegano enzimi speciali che riconoscono e correggono i nucleotidi appaiati impropriamente.
La nucleasi è l'enzima che che rimuove la porzione danneggiata, poi sostituita da altri nucleotidi, grazie alla polimerasi e la ligasi, in un processo chiamato riparazione per escissione dei nucleotidi.
La malattia ereditaria xeroderma pigmentoso è causata da un deficit di un enzima implicato nella riparazione per escissione, e comporta ipersensibilità alla luce e tumori alla pelle.
In linea di massima, la maggior parte dei processi di riparazione prevede l'intervento di molecole di DNA polimerasi.
Il normale replicamento di DNA non permette di completare l'estremità 5', e quindi cicli ripetuti di replicazione porterebbero alla produzione di molecole di DNA sempre più corte.
I telomeri sono speciali sequenze nucleotidiche, TTAGGGG, che non contengono geni.
Il DNA telomerico protegge i geni dell'organismo dall'erosione ed impedisce che le estremità attivino sistemi cellulari per monitorare il danno a carico del DNA.
La telomerasi è un particolare enzima che consente di ripristinare i telomeri accorciati, catalizzando il loro allungamento, grazie ad una molecola RNA che agisce come stampo per i nuovi segmenti del telomero all'estremità 3'.
La telomerasi non è presente nella maggior parte delle cellule pluricellulari, e quindi nelle cellule somatiche il DNA tende ad essere più corto nelle cellule degli anziani.
Quindi è probabile che i telomeri siano la causa della limitazione della durata della vita degli organismi.
La telomerasi è presente nelle cellule germinali che danno origine ai gamenti e quindi è presente nei neonati.
Purtroppo la telomerasi mantiene vive le cellule del cancro non facendo accorciare i telomeri.

Piaciuto l'articolo? Lascia un commento!
EmoticonEmoticon